Projekt zum Extrahieren der Daten der WISIA Webseite.
Hintergrund
Ich benötige die Daten der Artenschutzdatenbank des Bundesamt für Naturschutz in Bonn.
Deren Wissenschaftliches Informationssystem zum Internationalen Artenschutz liefert diese Informationen über eine Webseite. Weder API noch Datenbankkopie sind verfügbar.
Daher musste ich die Seite jeder einzelnen Art aufrufen, die Informationen extrahieren und verarbeiten.
Das Projekt
Mit dem Projekt speichere ich den Weg, wie ich meine Kopie der Artenschutzdatenbank aufgebaut habe. So kann jederzeit eine neue Version der Artenschutzdatenbank erstellt werden, beispielsweise nach einem Update der Original Datenbank.
Das Projekt ist weder universell, flexibel, konfigurierbar oder sonst was, sondern dient nur diesem einen Zweck.
Analyse
Auf der WISIA Seite kann man eine Recherche starten, zB zu "testudo":
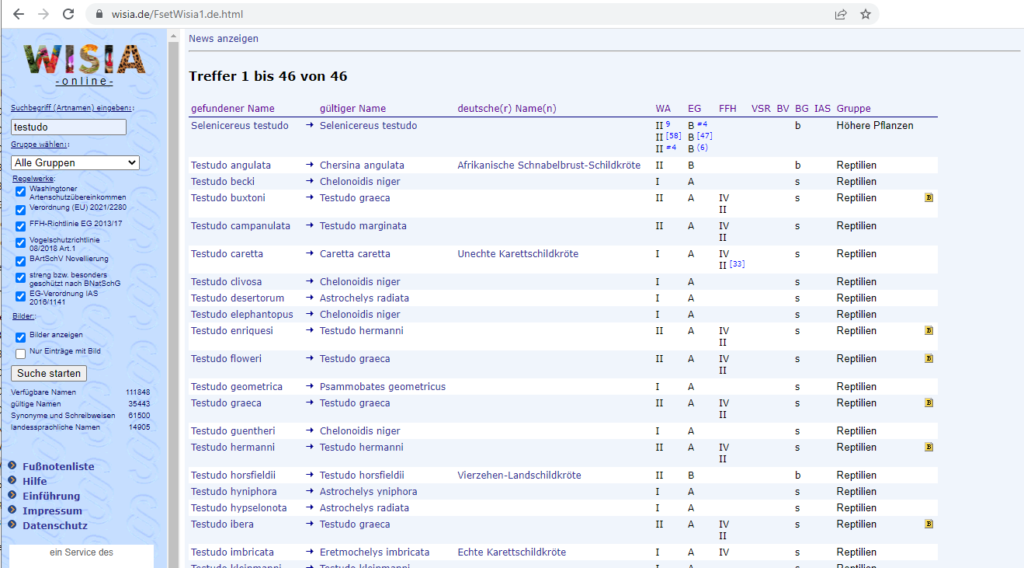
Von dort gelangt man auf die jeweilige Seite einer Art, zB "Testudo hermanni":
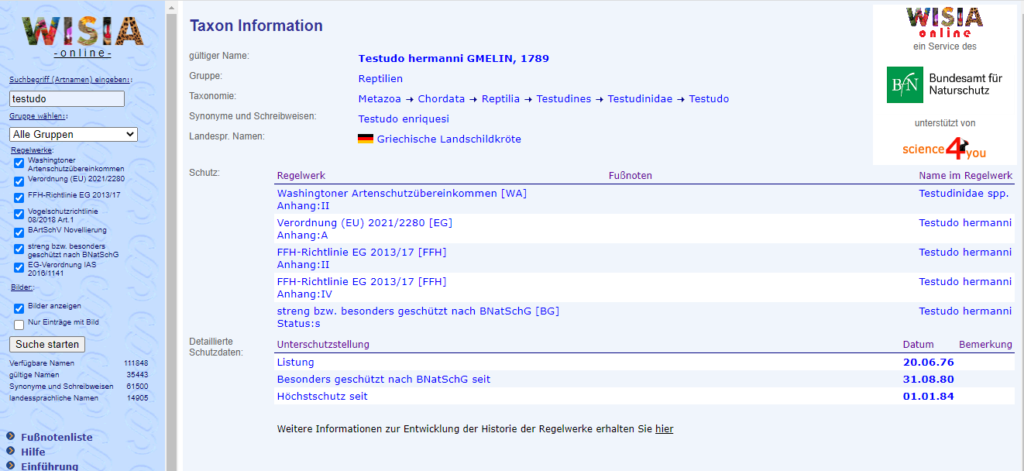
Eine Analyse des HTML Source Codes der Seite zeigt, dass die Seite über ein Frameset zusammengebaut ist und der rechte Teil mit den Informationen eine eigene Seite ist. Diese Seite wird über einen eindeutigen Parameter, der Knoten ID, aufgerufen.
Für Testudo hermanni lautet die Knoten ID: 19442.
Der Aufruf der Seite: https://www.wisia.de/GetTaxInfo?knoten_id=19442:
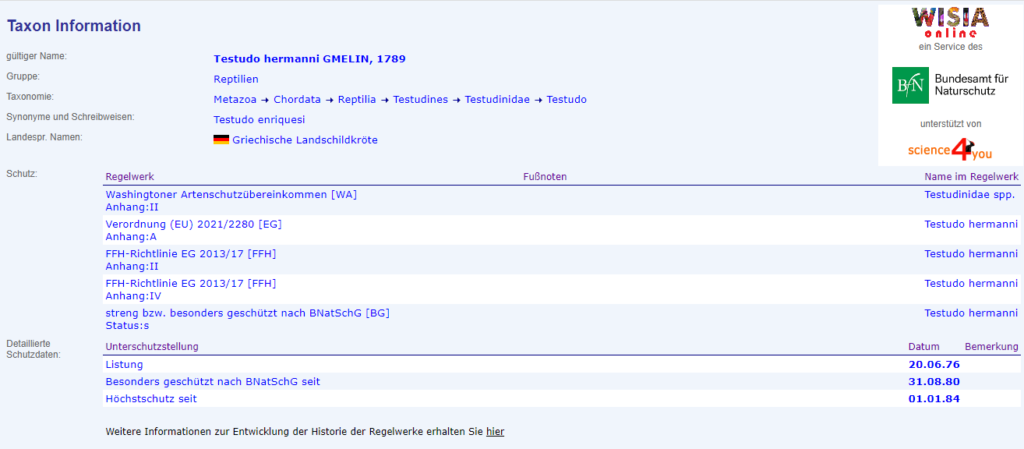
Auf der Seite finden sich strukturiert alle Informationen zu der Art.
Leider ist es technisch nicht so, dass es eine leere Vorlage der Seite ist, die anschließend zB über einen REST Aufruf, der ein JSON Objekt zurückgibt, gefüllt wird. Das hätte es mir bedeutend einfacher gemacht.
So muss ich für jede Art die komplette Seite laden und die Informationen selbst extrahieren.
Ablauf
TODO: funktionierendes Mermaid-Plugin installieren.
Workaround: Screenshot
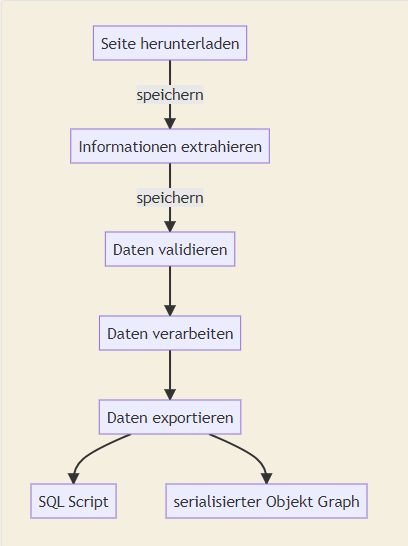
Seite herunterladen
In einem ersten Schritt lade ich die jeweilige Seite einer Art auf meinen Arbeitsrechner herunter und speichere sie. Anschließend kann ich mit der gespeicherten Seite weiter arbeiten und muss sie nicht jedesmal neu ziehen, um darauf zuzugreifen.
Die Seiten werden als GZIPte Objekte gespeichert.
Am Ende hatte ich über 7 GB gespeichert, bei einer Kompression auf ca. 25% habe ich also ca. 30 GB heruntergeladen.
Informationen extrahieren
Im nächsten Stritt werden die Informationen der Seite extrahiert und gespeichert.
Die Informationen werden als GZIPte Objekte gespeichert.
Die gespeicherten Objekte haben eine Größe von knapp 43 MB.
Für das Verarbeiten der Seite habe ich HtmlUnit verwendet.
<!-- https://mvnrepository.com/artifact/net.sourceforge.htmlunit/htmlunit --> <dependency> <groupId>net.sourceforge.htmlunit</groupId> <artifactId>htmlunit</artifactId> <version>2.70.0</version> </dependency>
Daten validieren
Seiten ohne gültigem Namen werden aussortiert, zu deren Knoten ID ist keine Art zugeordnet.
Seiten mit gültigem Namen aber ohne Taxonomie werden ebenfalls aussortiert.
Der gültige Name / wissenschaftliche Name ist nicht eindeutig, daher muss die eindeutige Knoten ID weiter verwendet werden.
Bei der Taxonomie bin ich davon ausgegangen, dass die Pfade alle eindeutig sind. Ich war überrascht, dass dem nicht so ist. Es existieren einige Einträge mit mehreren Elternknoten.
Beispielsweise Gomphus kann über Gomphidae, aber auch über Gomphaceae erreicht werden.
Das ist aber anscheinend korrekt, denn ich fand folgendes:
Duplicate name. This name, above species rank, is duplicated within the NCBI classification
https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?mode=Info&id=107809
Hier die uneindeutigen Taxonomien:
- Caryophyllidae : [Dicotyledonae, Scleractinia]
- Arenaria : [Scolopacidae, Caryophyllaceae]
- Cantharellus : [Fungiidae, Hygrophoraceae]
- Dracaena : [Teiidae, Agavaceae]
- Pholidota : [Mammalia, Orchidaceae]
- Gomphus : [Gomphidae, Gomphaceae]
- Mytilidae : [Mytiloida, Anisomyaria]
- Oenanthe : [Umbelliferae, Muscicapidae]
- Stelis : [Orchidaceae, Megachilidae]
Daten verarbeiten
Aus den extrahierten Daten muss ich eine Struktur ableiten und diese dann dorthin überführen.
Die verarbeiteten Daten werden als ein serialisiertes, gezippte Objekte gespeichert. Dieses ist weniger als 2MB groß und kann in wenigen Sekunden geladen werden.
Daten exportieren
Ursprünglich hatte ich die Idee, ein SQL-Schema zu schreiben und die Daten als INSERT-INTO-Scripte generieren zu lassen. Kann vielleicht nochmal kommen.
Letztendlich habe ich aber die Objekt Datei verwendet.